Escuchás un sonido repentino, fuerte, que te taladra la cabeza. No tenés idea qué es, dónde está, ni de dónde viene. Después de un segundo entendés: es la alarma. La peleás; ¿que tan importante es lo que tengo que hacer hoy? Decidís levantarte de la cama, vas para el baño y te mirás al espejo. Estás ahí, vos, todo lo que sos, todo lo que te hace ser y pensar lo que pensás. Entre decenas de ideas que cruzan tu cabeza, algunas sin respuesta, otras demasiado disparatadas para la hora, te detenés en una: ¿qué te diferencia de una bacteria o de una mosca?

No somos un virus, no somos un cobayo; tenemos algunas cosas en común y otras no.
La pregunta ‘¿qué nos hace ser quienes somos?’ es tan fundamental como antigua. Fue abordada de distintas maneras y a diferentes escalas, pasando por cosas como: somos bichos conscientes, podemos vivir en sociedad, hasta terminar en un ‘meh, es muy complejo’. Pero a alguien se le ocurrió ir a la base molecular del asunto. Al igual que el resto de los seres vivos conocidos, estamos hechos de muchas células que funcionan gracias a que tienen un manual de instrucciones en los genes. Dicho esto, la respuesta al dilema planteado parece obvia: si somos más complejos que una hormiga es porque tenemos más instrucciones que nos dan esta posibilidad; es decir, más genes.
A principios del siglo XXI, la secuenciación de genomas empezó a ser cosa seria. Se generaron muchísimos datos de muchísimas especies y la comparación entre ellas permitió sacar nuevas conclusiones. Entre ellas, que los Homo sapiens no tenemos más genes que la planta de arvejas. Sin embargo vieron que lo que sí tenemos, es una gran proporción de ‘ADN basura’.
Esta historia tiene que ver con entender que no todo ese material genético es basura, sino que puede tener funciones muy específicas y esenciales que hacen que seamos los organismos complejos que somos.
Perteneciste a una raza antigua
Históricamente se pensaba que la única manera de hacer efectiva la información del genoma (ADN) era a través de su ‘transcripción’ en moléculas que cumplían la función de llevar esa información a otros lugares y que a partir de estas se generaran las proteínas. En un principio, a estos paladines de la traducción de información, los ácidos ribonucléicos (ARNs), se los consideró únicos y con una sola función: ‘Todos los ARN que existen están involucrados directamente en la síntesis de proteínas’. Así fue como surgieron los luego llamados ARN mensajeros, nombrados así por su capacidad de transmitir (o codificar) la información del ADN para la formación de proteínas.
Sobre fines de los ‘90 se descubrieron en animales y luego en plantas unas moléculas de ARN distintas a las conocidas hasta el momento. La diferencia tiene que ver con que no contienen información que permite generar proteínas sino que cumplen un rol distinto, y se las agrupó como ARNs no codificantes.
Más adelante, a un subgrupo de estas moléculas (ARNs no codificantes) se los llamó microARNs, por su tamaño pequeño. Si un ARN mensajero −ese que lleva las instrucciones para hacer un proteína, como por ejemplo la queratina que forma las uñas− tiene unas 8600 unidades (nucleótidos) que lo conforman, un microARN tiene 21. Si un nucleótido fuera una cuadra, entonces el ARN mensajero de la queratina tendría unos 860 km −lo que equivale a la distancia de Rosario a Mendoza−, mientras que un microARN mediría un poco más de dos kilómetros.
Pero en este caso y como en otros asuntos de la vida, lo que nos interesa no es tanto el tamaño sino la capacidad de hacer. La función biológica de los microARNs en los organismos eucariotas (los que tienen células con núcleos) se asoció en un principio al control de cuándo y cuánto se expresan los genes que a su vez pueden influenciar la expresión de otros genes que pueden regular la expresión de otros genes, y así, en una sucesión similar a la cascada de señalizaciones que regula los intentos de relocalizar a una chiva que evidentemente no quiere salir de ahí. Esto no es menor, dado que a medida que te movés más y más hacia arriba en ese proceso, empezás a tocar la expresión de genes que puede determinar cuestiones más bien ontológicas, como la forma en la que se desarrollan los órganos o, en el reino vegetal, determinar la forma de las hojas, que las plantas florezcan (y cuándo) o que puedan crecer normalmente y completar su ciclo de vida. Después de mucha investigación se llegó a la conclusión de que los microARNs funcionan como interruptores en estos procesos.
De ADN basura a reguladores de cuestiones ontológicas. Parece que de entrada le erramos por casi nada.
Modelo para armar
Si bien los microARNs son pequeños, se encuentran contenidos dentro de un ARN de mayor tamaño, un precursor que los porta. El truco está en que el microARN dentro del precursor no es capaz de prender ni apagar la expresión de ningún gen; no es funcional, sólo actúa como interruptor de genes una vez que es cortado y liberado. Esto se denomina procesamiento. Sí, una vez bautizamos a un proceso con un nombre más bien claro y descriptivo.
En el laboratorio de Biología del ARN perteneciente al Instituto de Biología Molecular y Celular de Rosario tratamos de entender cuáles son los indicios dentro del precursor que revelan dónde está el microARN. Es como una búsqueda del tesoro, sólo que el mapa mide 0,00000021 metros, el tamaño aproximado de una de estas moléculas.
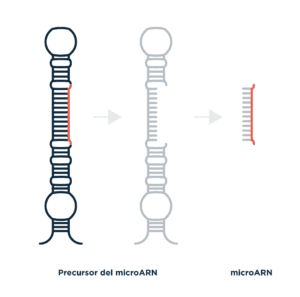
Existe una enzima, llamada DICER, que es la encargada de leer el mapa del tesoro en el precursor y liberar el microARN. Funciona como el gigante Procusto de la mitología griega, un posadero que ofrecía su casa a los viajeros. Sin embargo, Procu (?) tenía una única cama exageradamente pequeña y, si el viajero era alto y su cuerpo era más largo que la cama, cortaba sus extremidades mientras este dormía. DICER funciona de una manera similar: reconoce al microARN dentro del precursor y se deshace de todo lo demás. A su vez, un mismo tipo de microARN puede estar contenido en diferentes precursores. Cada uno de estos precursores constituye un miembro de la misma familia.

Procusto corta piernas
Dime cómo te procesas y te diré quién eres
Este procesamiento ocurre tanto en plantas como en animales, incluidos seres humanos (sí, todos tenemos microARNs y DICER adentro). La diferencia está en el número de precursores distintos que pueden tener, diferenciados entre sí por la forma y el mecanismo de procesamiento que da lugar al microARN maduro. Estas diferencias entre plantas y animales sugieren que los microARNs aparecieron de manera independiente a lo largo de la evolución en ambos reinos (a esto se lo llama evolución convergente).
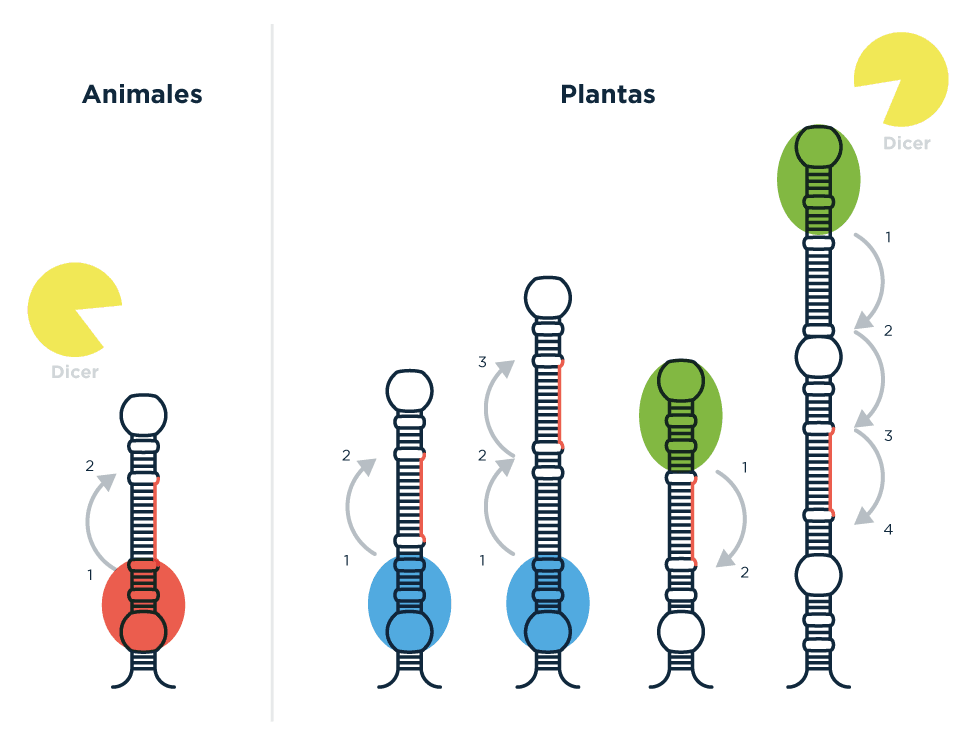
Mecanismos de procesamiento en animales y plantas y reconocimiento histórico al PacMan como influencia visual inescapable. Los números indican el orden y la dirección en la que DICER corta los distintos precursores.
La gran mayoría de los trabajos en plantas sobre este tema fueron realizados en Arabidopsis thaliana, un modelo vegetal muy usado debido a su pequeño tamaño y su corto ciclo de vida, pero de poca utilidad para la producción alimenticia.

Mamífero modelo, planta modelo, gusano modelo, modelo modelo.
Desde el 2013 en el laboratorio comenzamos a preguntarnos cómo sería el procesamiento de microARNs en otras especies vegetales como soja, papa, tomate, maíz y arroz, entre otras. Queríamos saber si las reglas que se habían descrito para Arabidopsis eran válidas en especies de interés agronómico.
Nos basamos en la idea de que, si a lo largo de miles y miles de años de evolución las reglas del procesamiento de microARNs se habían mantenido, podrían encontrarse los mismos indicios moleculares en diferentes precursores sin importar qué tan distintos sean, por ejemplo, la papa de Arabidopsis.
Para responder este interrogante empleamos herramientas bioinformáticas junto con la generación de plantas transgénicas. La ventaja de entender cómo funciona algo es que podemos hacer más y mejores cosas; por ejemplo, que una planta exprese un microARN bajo determinadas condiciones que uno elige. Es como jugar a ‘Simón dice’ y hacer que la planta florezca independientemente de las señales externas que reciba, como la cantidad de horas de luz o la temperatura.

De fenómeno a fenotipo hay un solo microARN. Al menos eso Dicer.
¿Cómo se estudia algo que no se ve? Una de las maneras es aumentando los niveles de uno de los precursores de microARN. Pero además se pueden hacer versiones mutantes del mismo, es decir, generar plantas transgénicas que expresen dos precursores distintos: uno mutante y uno tal como está en la naturaleza. Una de las cosas más lindas de hacer ciencia es que se pueden crear cosas que antes no existían en la naturaleza y usarlas para responder preguntas.
Las plantas transgénicas con niveles aumentados de microARNs muchas veces son muy diferentes de las plantas silvestres. Lo bueno es que se suele trabajar con caracteres distinguibles a simple vista. A las células de plantas transgénicas podemos ‘obligarlas’ a generar precursores truncados (interrumpidos), es decir, que no son funcionales. Esto nos permite diferenciar si la porción que se recortó es importante para el procesamiento en función de si obtenemos plantas que se parecen más a una planta silvestre que a la planta que sobre-expresaba el precursor original. Al mutar la secuencia del precursor, generando la versión truncada, DICER no ‘sabe’ dónde hacer los cortes en los precursores, por lo que los microARNs nunca se liberan y por lo tanto no son funcionales. En palabras (todavía) más técnicas: no se ve modificado el fenotipo de las plantas (no lucen distintas).
A continuación viene una segunda etapa de caracterización molecular para tratar de entender un poco mejor qué está pasando con los microARN que no están generando ningún cambio visible en las plantas. Lo primero es asegurar que el precursor se está expresando a montones en las células; y si esto es así, ¿el microARN se está acumulando o se degrada? Para esto último se usa una técnica llamada Northern Blot, donde se visualiza indirectamente el microARN a través de una molécula de ADN radiactiva que lo reconoce específicamente y permite que se pueda ver por revelado fotográfico.
En última instancia, el trabajo del biólogo molecular es hacer visible lo invisible. Parece ser que la clásica idea de ‘la información que está en el ADN se transcribe en ARN y de ahí se traduce en forma de proteína’ no es tan así, sino que en realidad es algo como ‘la información que está en el ADN puede o no transcribirse a ARN, que puede o no codificar para proteínas, o también puede ser una señal tipo microARN, que puede no procesarse o acumularse’. Lo que, una vez más, nos muestra que la biología es hermosamente rica y compleja.
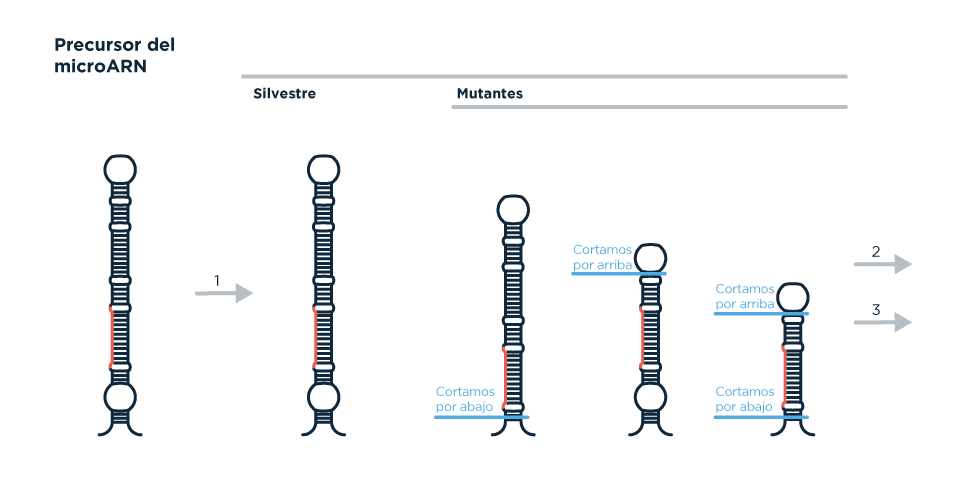
(1) Hacemos mutaciones sobre el precursor. (2) Generamos plantas transgénicas con niveles aumentados del precursor. (3) Evaluamos los fenotipos de las plantas transgénicas.

(4) medimos las cantidades de microARN maduro mediante Northern Blot usando una molécula de ADN radioactiva que lo reconoce específicamente.
Por suerte, existen grupos de investigación que secuencian genomas y los depositan en bases de datos. Eso nos brinda las secuencias y ubicación de los microARNs, pero no la información sobre su procesamiento. Teníamos el qué y el dónde, pero no el cómo. Para eso, usamos herramientas bioinformáticas que simplifican este trabajo, destacan lo importante y descartan lo que no interesa.
Partiendo de muchos microARNs conocidos previamente y estudiados para la ocasión, lo que hacemos −de la mano de nuestro bioinformático amigo− es predecir cuáles son los precursores que mantienen la misma función a lo largo de la evolución en esas otras especies de plantas más interesantes y atractivas que Arabidopsis(conocidos como genes ortólogos). Hagamos algunos números: en Arabidopsis hay 29 familias de microARNs conservados en plantas que están codificados por 96 precursores distintos; cuando agregamos a los precursores ortólogos sumamos 2112 precursores para analizar, un número no analizable a ojo.
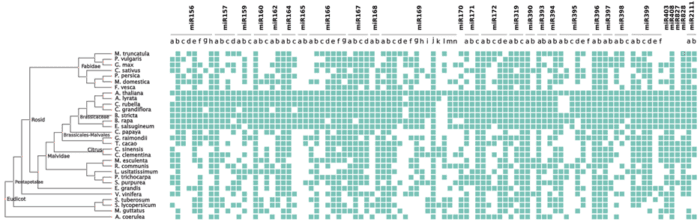
Identificación y conservación evolutiva de los precursores de microARNs de plantas. Sí, cada cuadradito verde es un microARN diferente.
La manera de entender los datos es alinear sus secuencias y compararlas para encontrar patrones comunes entre ellas, mirar qué cosas son similares y qué cosas no. Además, se hacen gráficos para poder representar toda esta información en distintas especies de plantas al mismo tiempo. Estos gráficos permiten ver diferencias y similitudes a simple vista, sin tener que repasar uno a uno los datos, y detectar qué parte de los precursores de microARNs están conservadas en las distintas especies. <3 DATA VIZ <3
Representamos los precursores de microARNs empleando un software llamado Circos, que permite generar gráficos para la visualización de datos e información. Los datos son representados de manera circular, lo que hace que los Circos sean ideales para explorar las relaciones entre los objetos, posiciones o los personajes de LOST.
Podemos ver las relaciones entre cada personaje. Las líneas naranja unen familiares, las rojas unen amoríos, la línea ausente es la argumental donde nos dicen qué onda el oso polar y otras cosas que solucionaron en un final bastante poco consistente. #NotPennysBoat
Analizando muchos CIRCOs encontramos huellas evolutivas conservadas en el procesamiento de los precursores de microARNs, lo que nos permitió tener una visión integradora del procesamiento de microARNs en más de 30 especies vegetales diferentes.
A su vez, tener una visión global de todas las especies más allá de Arabidopsis nos permitió identificar nuevos mecanismos de procesamiento, pistas aún ni siquiera descriptas. Esta vez la novedad vino de la mano de un microARN conservado a lo largo de la evolución, el miR166, que es responsable del correcto desarrollo del tallo de las plantas, y que determina que la parte de arriba y abajo de las hojas sean diferentes. El procesamiento de este microARN es completamente distinto a todo lo que sabíamos, y recién estamos comenzando a juntar las piezas de su rompecabezas molecular. Una de las formas en las que pudimos saber que era distinto fue viéndolo hacer cosas que otros microARNs no podían.
Entender el procesamiento de los microARNs y el rol que cumplen en especies vegetales de interés agronómico es el primer acercamiento a potenciar su capacidad como producto alimenticio.
Si bien estos descubrimientos fueron interesantes en sí mismos, no dejan de ser un qué, cuando una parte divertida del trabajo y en la que queremos hacer énfasis es en el cómo. Para que esto sucediera se requirió un ida y vuelta entre personas y máquinas, dinámica que implicaba que cada parte hiciera lo que mejor hace. Nosotros diseñamos el experimento y la lógica básica, pero los volúmenes de información eran demasiado grandes, así que le pedimos a una computadora que ordenara y presentara esa información de una manera que pudiéramos abordar. Así, la información volvió a nosotros en formas visuales que nos permitieron entenderla y masticarla aún cuando era muchísima.
Empezamos hablando de hacer visible lo invisible, y eso no se limita a revelar una secuencia de ARN o fotografiar una proteína fluorescente, sino también a usar informática para zarandear pajares y encontrar las historias que cuentan las agujas. Usamos luz ultravioleta y microscopios para ver lo que nuestros ojos, pero también software para convertir tablas infinitas que nuestras cabezas no pueden abarcar en imágenes que un cerebro altamente visual entienda. Expandimos nuestra capacidad para poder hacer preguntas nuevas y convertimos en parte de nuestra cotidianeidad herramientas que hace 100 años hubiesen conmovido a cualquier científico.
Esta nota está basada en un trabajo publicado en mayo de este año por el grupo de Biología del ARN del Instituto de Biología Molecular y Celular de Rosario.





