—Mirá, tiene los ojos de papá.
—Sí, y los rulos de mamá.
—Aunque la nariz la recibió de la nona.
Todos escuchamos un diálogo parecido a ese alguna vez, probablemente porque mucha gente sabe que los rasgos físicos se heredan, a veces de forma más clara, a veces menos, siempre atravesadas por el ambiente en el que el organismo se desarrolla. Así, algunas de las cosas (no todas) que nos hacen ser quienes somos están también inscriptas en nuestro ADN, esa famosa doble hélice, si es que existen hélices famosas.
Desde hace mucho sabemos dos cosas fundamentales sobre el ADN: que funciona como un manual de instrucciones, es decir, una maraña de información que permite a las células (o a los virus) ser como son. Y también que pasa de progenitores a progenie; se hereda, haciendo que el maestro diga ‘tenés los ojos de tu papá’. No, señor, disculpe que me ponga técnico pero no tengo sus ojos, más bien estoy expresando genes que vinieron en un espermatozoide de papá que tuvo más éxito que el resto en encontrar otra mitad de genoma (y un montón de reguladores de la expresión genética que mucho más tienen que ver con mamá que con él), y multiplicarse hasta formar estos ojazos.

Señor McClure, ¿qué significa ADN?
El ADN fue una de las moléculas superestrellas durante gran parte de los últimos dos siglos. En ese lapso pasamos de postular la existencia de una partícula que se transmitía de generación en generación a entender algunas de sus funciones, y también a saber muchísimo sobre su estructura. Tanto que con el tiempo fuimos madurando la idea de que ese manual de instrucciones estaba escrito en códigos. Y eso cambió todo. Si sabemos que hay enfermedades que pasan de progenitores a hijos, ¿por qué no pensar que están en el ADN? Si tan sólo existiera alguna manera de descifrar ese código, de leerlo y entender qué significa.
En 1990, un grupo de científicos trabajando en Estados Unidos se empezó a preguntar cómo sería el genoma de los humanos, es decir, cuál y cómo es la lista completa (o casi completa) de genes (y otra información que no necesariamente es transcripta o traducida, pero que es recontra importante) presentes en el ADN humano. Querían ver qué información en nuestras células es la que nos hace ser como somos y no como cualquier otra cosa viva.
Empezaron por lo que todos los científicos hacen cuando empiezan un proyecto: ̶i̶d̶e̶a̶r̶ ̶u̶n̶ ̶p̶l̶a̶n̶ ̶d̶e̶ ̶t̶r̶a̶b̶ buscar subsidios. Después de un poco de convencimiento, lograron que el NIH (National Institutes of Health – Institutos Nacionales de Salud de Estados Unidos) y el Departamento de Energía de Estados Unidos les dieran poquita plata ―unos 3 mil millones de dólares― y entonces armaron un plan para leer e interpretar este manual de instrucciones que todos llevamos dentro. Lo que ocurrió luego fue fascinante. Intrigas, renuncias, competencias. Resulta que…
¡Momento! Primero, un cachito de historia
El ADN es una enorme cadena formada por una secuencia de unidades parecidas (llamadas nucleótidos) que pueden diferir en una de sus partes. Así, cada pieza de la cadena de ADN puede contener una base nitrogenada distinta que simbolizamos con las letras A, C, G y T (existen más pero, en principio, vamos con esas). Entonces podemos decir que se forma una secuencia en un alfabeto de sólo 4 letras. Y como es una cadena, el orden de estas letras define qué se encuentra ‘escrito’ en esa secuencia, de qué gen se trata.
Lo cierto es que a mediados del siglo XX ya se sabía que esta secuencia era un componente clave en todos los seres vivos y los virus, pero el problema era leerla. Un poco más adelante, durante la década del ‘70, un científico llamado Frederick Sanger, que ya venía con la idea de resolver enigmas relacionados con otras ‘letras’, las de las proteínas (razón por la cual ganó su primer Nobel), desarrolló un método para poder secuenciar el ADN y conocer qué letras (nucleótidos) y en qué orden formaban parte de una molécula de ADN. Y con esa técnica en 1977 secuenció el genoma de un virus, el primer ácido nucleico totalmente secuenciado de la historia, con una longitud de 5370 ‘letras’ o nucleótidos.
El rompecabezas de Sanger.
Su método consiste, literalmente, en armar un rompecabezas pieza por pieza. Pero esta técnica tenía un problema: permitía leer hasta mil y algo de letras por vez, mientras que el genoma humano tiene unas 6 mil millones. Pequeño limitante. La parte experimental era muy lenta, cara y un poco difícil de hacer. Pero de todos modos, esta primera versión sirvió mucho para empezar a leer secuencias de genes cortos, formados por pocas letras.
Con cada pequeño avance la curiosidad crecía. ¿Qué descubriríamos si pudiésemos conocer la totalidad de la secuencia del genoma humano? ¿Sabríamos con exactitud qué nos vuelve humanos? ¿Tenemos más genes que otros organismos? ¿Podemos conocer la causa de enfermedades genéticas? Todas esas preguntas y más se hizo un grupo de gente hacia fines de los ‘80 mientras conseguían los mencionados subsidios que, por cierto, no vinieron de forma muy desinteresada: el NIH quería indagar de qué manera nuestro genoma podría ayudar a avanzar a la medicina, mientras que el Departamento de Energía pretendía conocer qué efectos podrían causar el uso de la radiación y la energía atómica al ADN humano.
A modo de prueba empezaron por genomas que se sabía que eran más chicos: en 1995 el de algunas bacterias o en 1996 el de la levadura de la cerveza. Pero eventualmente había que dar el gran paso, y al igual que en los otros organismos, la metodología para secuenciar el genoma humano era simple pero laboriosa. Consistía en extraer ADN de las células de donantes anónimos y fragmentarlo en pedazos más chicos. Cada institución partícipe del proyecto recibía varios de estos fragmentos ordenados que debía fragmentar aún más (y al azar) para obtener ‘regiones legibles’, o sea, de aproximadamente mil ‘letras’, porque era la única manera en la que podían ser leídas por la tecnología que Sanger había desarrollado. Así, secuenciando muchísimos fragmentos que se superponen entre ellos, se podía reconstruir la molécula original.
Dicho de otro modo: la maquinaria que había en ese entonces permitía sólo secuenciar unos pocos fragmentos en un par de días, por lo que la única forma de secuenciar millones de fragmentos era tener muchas máquinas paralelas trabajando continuamente y después juntar todo.

Como un ciber pero mejor.
En ese entonces Craig Venter, un investigador del NIH que trabajaba en bacterias, había estado desarrollando un método más eficiente para secuenciar ADN, conocido como ‘shotgun sequencing’ (algo así como ‘secuenciación a escopetazos’, o más en criollo: ‘secuenciación al voleo’), que proponía leer miles de fragmentos simultáneamente sin un orden particular. Si bien a simple vista esto puede sonar extraño, lo cierto es que en 1995 Craig logró secuenciar el genoma de una bacteria en tiempo récord. Esto hizo que pisara fuerte en la secuenciación del genoma humano y comenzó una lucha de egos con el director del Proyecto Genoma Humano: James Watson, ese que ganó el Nobel por descubrir la estructura del ADN (en 1953) y siempre se caracterizó por ser difícil de tratar.
La relación entre dos tipos tan intensos no terminó nada bien y, en 1999, Venter terminó despidiéndose del NIH y fundó Celera Genomics, su propia compañía que comenzó su propio proyecto del genoma humano pero con fondos privados.

“Formaré mi propio ‘Proyecto Genoma Humano’ con fondos privados”. Interpretación laxa y no oficial de lo sucedido.
En esos años, muchos investigadores que descubrían genes los patentaban; eso hacía que fueran los únicos habilitados a seguir investigando esos genes, barriendo con la competencia. Y esto abrió la puerta a debates acalorados en la ciencia. ¿Alguien podría ser ‘dueño’ del genoma de nuestra especie? ¿Se puede patentar el genoma humano? La ida de Venter a una empresa y el tema del patentamiento hizo que comenzara una carrera entre los proyectos público y privado sobre quién sería el primero en tener la secuencia completa del genoma humano.
La disputa terminó cerca del año 2000, cuando el presidente de Estados Unidos, Bill Clinton, decidió ponerse en el medio y lograr un acuerdo. En junio de ese año dio una conferencia de prensa junto a Venter y Francis Collins (sucesor de Watson en el proyecto público) anunciando la publicación de el primer borrador del genoma humano público y gratuito, accesible por todo el mundo.

“Entonces, uno quería secuenciar todo ordenadito, y el otro todo al voleo, y les dije, che, júntense, y pim pam pum, acá estamos, todos sonriendo y con la cosa secuenciada.” (más declaraciones no oficiales, tirando a inventadas). Craig Venter, Bill Clinton y Francis Collins en la Casa Blanca el 26 de Junio del 2000. Foto: Ron Sachs
Al año siguiente, ambos proyectos fueron publicados en las dos prestigiosas revistas científicas Nature (proyecto público) y Science (proyecto privado).
El impacto
Con esto se abrió una puerta de posibilidades al entendimiento de nosotros mismos pero también de muchísimas otras especies. Por ejemplo, gracias a la secuenciación del genoma humano (Homo sapiens) y del genoma del neandertal (Homo neanderthalensis), aprendimos que nuestras especies se separaron hace aproximadamente medio millón de años y luego, cuando se volvieron a cruzar, lograron que ahora casi todos los humanos tengamos una pizca de ADN neandertal en nuestro genoma.
Pero quizá la rama donde puede tener mayor impacto es la aplicación de estudios genómicos en pacientes. Hasta ahora gran parte de la medicina se manejó con conceptos derivados de la estadística: ‘a la mayoría de las personas con cierta condición, les sirve tal medicamento, entonces a cada paciente nuevo que aparezca con los mismos síntomas se le receta esa misma droga o tratamiento’. El problema es que hay una gran cantidad de casos en los que eso no funciona porque, claro, estadística. Pero si la enfermedad fuera causada únicamente por algo genético y lográsemos conocer del genoma los pedacitos relevantes, sería posible tener un marcador claro y preciso que permitiría tomar mejores decisiones en cuanto a los tratamientos a recetar. Ya no en términos estadísticos, sino personalizados y dirigidos.
Ya pasó mucho tiempo desde la secuenciación del primer genoma humano en el 2001. Nuevas tecnologías permitieron que los costos de secuenciar un genoma disminuyan considerablemente. El primero costó muchas pero muchas horas de trabajo y varios miles de millones de dólares, pero hoy se puede secuenciar un genoma por pocos miles de dólares y la tendencia indica que el precio seguirá bajando. Tanto que, si sigue así, puede ser que en el futuro cercano secuenciar un genoma se convierta en un protocolo tan fácil como cualquier chequeo de rutina.
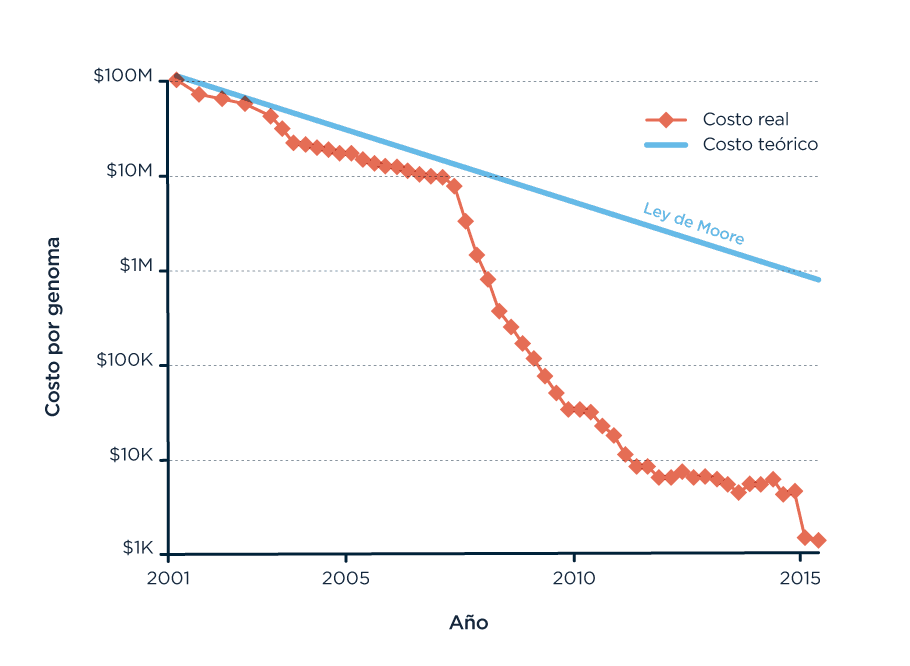
Comparación de la proyección del costo teórico con el costo real de secuenciar un genoma. La ley de Moore, mal y pronto, es una proyección de velocidad de avance de la tecnología. Donde se rompe la linealidad, ahí encontramos una disrupción tecnológica, como fueron las técnicas de secuenciación de segunda (principios de 2008) y tercera generación (hace 3 o 4 años). Fuente.
Esta reducción sustancial en los costos permite que se abran nuevas líneas de investigación y aplicación de las tecnologías en la medicina actual. Existen cuatro grandes áreas que están en auge en el uso de información genómica, en esta nueva área conocida como Genómica Médica:
La primera es la Genómica del Cáncer, y vale en este punto recordar que el cáncer es (también) una enfermedad del genoma. Una serie de cambios pueden transformar una célula normal en una célula tumoral, que crece y se divide sin control, creando masas tumorales en el cuerpo. Conocer qué mutaciones posee un tumor podría permitir orientar el tratamiento para combatirlo. Cabe aclarar que esto sólo funcionaría en determinados tipos de tumores, generalmente los menos agresivos; ya que los tumores más agresivos suelen estar compuestos por una diversidad de poblaciones de células no despreciable (que nace precisamente de la alta tasa de mutación de su genoma).
La segunda es la Farmacogenómica, porque nos estamos dando cuenta de que la forma en la que se administran medicaciones es ampliamente imprecisa. Las personas pueden responder distinto a ciertas drogas y una de las causas puede ser porque su genoma es distinto. Así, una droga que a cierta persona puede serle beneficiosa, a otra le resulta ineficaz. Hoy en día el 7% de las drogas aprobadas por la FDA (la Agencia de Drogas y Alimentos de Estados Unidos) tiene información que puede ayudar a establecer las dosis indicadas según el genoma del paciente.
También se abre la posibilidad para el diagnóstico de enfermedades poco frecuentes. Se considera que una enfermedad poco frecuente es aquella que afecta a menos de 1 persona entre 2000. Sabiendo esto, y que se conocen alrededor de 8000 enfermedades poco frecuentes distintas, el resultado es que en conjunto afectan aproximadamente a un 7% de la población mundial. Sólo en Argentina, se estima que 3,2 millones de personas sufren una de estas enfermedades a lo largo de su vida. Muchas de estas familias con miembros afectados pasan años intentando conseguir un diagnóstico certero que explique su aflicción (en promedio hasta 7 años), sin contar los numerosos profesionales de la salud que visitan. La genómica puede ayudar a diagnosticar tempranamente y en tiempos más cortos a algunos de estos pacientes, pudiendo así ayudar a las familias de forma dirigida para su tratamiento.
La última, con ribetes éticos interesantísimos, es la Genómica en el embarazo. En Argentina se realizan con obligatoriedad estudios neonatales que intentan identificar a aquellos bebés con alguna de 5 enfermedades congénitas raras (Fenilcetonuria, Galactosemia, Deficiencia de Biotinidasa, Hipotiroidismo congénito primario e Hiperplasia suprarrenal congénita) que, de detectarse y tratarse tempranamente, se pueden revertir. Actualmente se está contemplando realizar estudios prenatales genómicos para detectar numerosas enfermedades en un solo ensayo y de forma no invasiva: sólo tomando un poco de sangre de la madre, sin tocar al feto en lo absoluto. Además, todo el tiempo nacen bebés agudamente enfermos, y muchas veces mueren al poco tiempo por no tener un diagnóstico temprano (en Argentina, en el año 2016, por ejemplo, fallecieron 3.027 recién nacidos durante la primera semana de vida). La secuenciación y detección temprana de enfermedades genéticas podría ayudar a salvar algunas de esas vidas.
Si bien parece de ciencia ficción, es algo que se está empezando a implementar. En Argentina se comenzaron a incubar los primeros proyectos de medicina personalizada en pacientes con enfermedades congénitas poco frecuentes. De hecho existe una manera un poco más barata de hacer esto que no tiene tanto que ver con secuenciar un genoma entero sino con ver si determinados pedacitos de genoma se encuentran en las células de una persona. Se les pudieron acercar estas nuevas tecnologías de secuenciación a pacientes con enfermedades genéticas que nunca tuvieron un diagnóstico claro. Se les pudo decir ‘lo que tenés −o lo que tiene tu hijo/a− es tal enfermedad’. Ya de por sí, exista o no un tratamiento disponible, esto significa un gran avance y un enorme peso que se le quita al paciente y a sus familiares.
No sin cierta elegancia, saber cada vez más sobre los genomas nos hizo dar cuenta de que también hay muchísimas cosas que no se pueden explicar mirando sólo la secuencia de ADN que nos hace, reforzando una vez más la idea de que es necesario mirar el contexto en el que se encuentran esos genomas, pero al mismo tiempo abrazando la necesidad de no negar las diferencias en esa larga cadena que le da al individuo su punto de partida. Todavía se sigue investigando no sólo qué función cumple cada parte de nuestro genoma, sino cómo interacciona cada una de estas partes con aquello que nos rodea. Los genomas no son todo, pero quizá sí son esa primera pieza que nos orienta y nos permite armar cada vez mejor este rompecabezas que llamamos vida.





